GIMS.dx
Was ist GIMS.dx?
GIMS.dx ist eine Plattform für die Prozessoptimierung jeglicher Art von NGS-basierter Diagnostik - sowohl für Keimbahn- als auch für somatische Mutationen. Dies beinhaltet die Diagnostik seltener Erkrankungen, die Identifizierung onkologischer Prädispositionsfaktoren als auch die Analyse von Tumormaterial.
GIMS.dx bietet Ärzten und Wissenschaftlern in der Genetik und Pathologie strukturierte und stufenweise Workflows für die Entscheidungsfindung und Dokumentation von Variantenklassifikationen, klinischen Interpretationen und Empfehlungen.
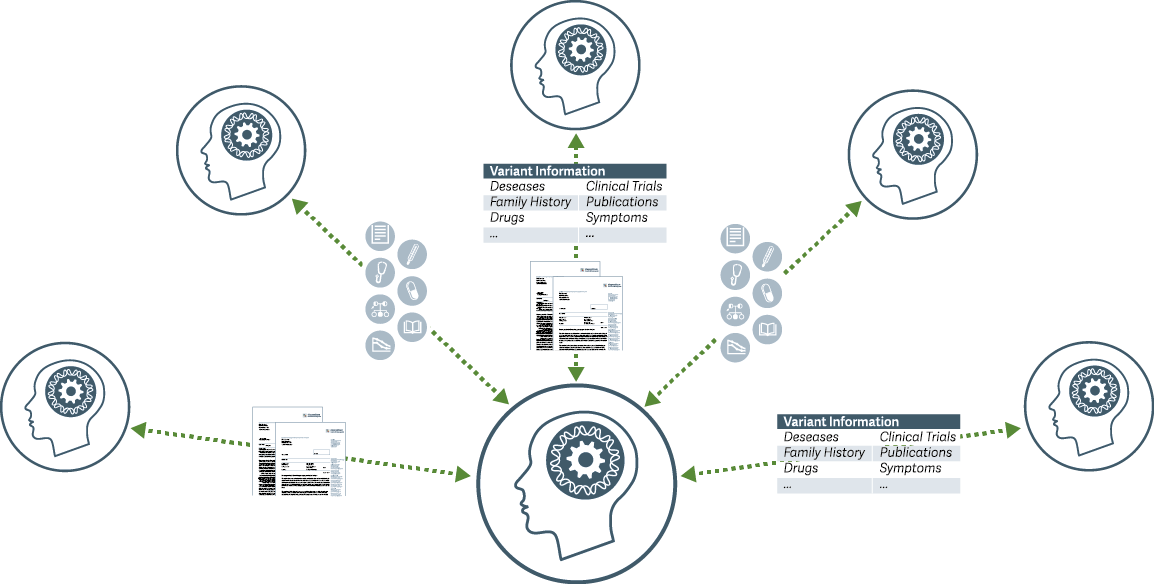
Durch die Nutzung von GIMS arbeiten molekulargentische und klinische Teams in einem digitalen genetischen Ökosystem zusammen. Aggregiertes Wissen und Entscheidungen werden transparent und damit vertrauenswürdig. Das faire Teilen von Varianteninformationen, genetischem Experten-Knowhow und klinischen Beobachtungen ist Teil eines kontinuierlichen Wahrheitsfindungsprozesses.
Wie es funktioniert

1. Variantenliste wird hochgeladen - manuell oder über Schnittstellen
2. entdeckte Varianten werden gelistet und automatisch mit Metadaten aus öffentlichen Quellen angereichert
a) ist die Variante bereits bekannt und ausreichend bewertet, wird sie automatisch prozessiert und für die Erstellung eines klinischen Befundes herangezogen
b) für die Prozessierung noch nicht klassifizierter Varianten wird der Nutzer schrittweise durch einen Variantenbewertungs- und Interpretationsprozess geleitet; Entscheidungen und Argumentationen können dokumentiert und versioniert werden
c) falls vorhanden, kann der Nutzer Informationen und bestehende Bewertungen aus dem Netzwerk beziehen
3. abgeschlossene Bewertungen können mit flexiblen Workflows geprüft und freigegeben werden
a) sobald die Bewertung einen gewissen Reifegrad erreicht hat, kann der Nutzer entscheiden, diese mit dem Netzwerk zu teilen
4. Interpretationen, Diagnosen und Empfehlungen werden in einem klinischen Befundbericht zusammengefasst und wo sinnvoll mit den Kommentaren des Nutzers ergänzt
5. Ergebnisse können
a) als druckfertige Dokumente geliefert werden
b) in EHR-Systeme integiert werden
c) über flexible und sichere digitale Kanäle verteilt werden
die wichtigsten vorteile
Schrittweise Führung durch Variantenklassifikations- und Interpretationsprozess:
- identifizierte Varianten werden automatisch mit verfügbaren Metadaten aus öffentliche Quellen ausgestattet, wie Referenzgenom, Gen-Nr., Transkriptnr., Chromosom, rs-Nr., Varianteninformation)
- erhöhte Produktivität, Zeitreduktion für Variantenanalyse
Strukturierter Dokumentationsprozess mit klaren Workflows für Prüfung und Freigabe:
- macht Entscheidungsprozess transparent und nachvollziehbar - ideal auch für Qualitätssicherung und Audits
- liefert kuratierte Informationen für diagnostische Entscheidungsunterstützung
- erlaubt strukturierten Aufbau einer eigenen Wissensbasis
- unterstützt automatisierte Erstellung diagnostischer Befundberichte
- ermöglicht Zeitersparnis und sichert Standardisierung sowie konstante Qualität des diagnostischen Outputs
- ermöglicht das Teilen von Inhalten und Wissen im Rahmen eines wachsenden Netzwerks von Laboren, wissenschaftlichen Institutionen und Expertengruppen
Contact block
Durch Ausfüllen und Versenden des Kontaktformulars stimmen Sie automatisch der Verarbeitung Ihrer Daten gemäß Artikel 6, Absatz 1a der DSGVO zum Zweck der Beantwortung Ihrer Anfrage zu. Weitere Informationen finden Sie in unserer Datenschutzerklärung.
